发布时间:2023-09-05
近日,中国科学院广州生物医药与健康研究院与澳门大学合作在Cell Research在线发表题为Structural basis of nucleosome deacetylation and DNA linker tightening by Rpd3S histone deacetylase complex的研究论文。该研究通过生化手段及单颗粒冷冻电镜技术确定了Rpd3S组装模式,并且以多种不同核小体底物模拟Rpd3S去乙酰化过程中的不同状态,成功捕获了Rpd3S在H3K36甲基化依赖的去乙酰化过程中的多个构象,以及与Linker Histone H1共存的模式。基于以上结果本研究提出:Rpd3S通过其Eaf3亚基上的CHD识别H3K36me3,并利用Sin3 basic surface与DNA的静电相互作用作为锚点,以多个不同的模式与核小体底物相结合,来移除不同区域组蛋白尾巴赖氨酸的乙酰基;另外,Rpd3S完成去乙酰化功能伴随着双侧DNA linker α角度的减小,提示Rpd3S不但可以擦除带负电的乙酰基团,同时也可能以与linker DNA 直接作用的方式来收紧DNA并帮助压缩染色质;而与H1的共存模式进一步提示了去乙酰化复合物与连接组蛋协同凝缩染色质的可能性。
在真核细胞中,组蛋白去乙酰化酶(Histone deacetylation, HDAC)以依赖于上游组蛋白修饰的形式来调控基因的转录水平,同时防止隐性转录的发生。在酿酒酵母(Saccharomyces cerevisiae)中,组蛋白去乙酰化酶Sin3 HDAC以Rpd3S和Rpd3L两种多亚基复合物形式分别存在于基因编码区及启动子区域。在基因转录过程中,Set2-Rpd3S通过联系组蛋白甲基化状态和去乙酰化的进程来维持染色质的稳定性,并防止异常隐性转录的发生。在过去的报道中,Rpd3S被认为不仅可以对所有四个组蛋白尾巴上的特定赖氨酸乙酰基发挥作用,并且能同时稳定核小体的动态变化。然而,Rpd3S多位点、多功能性的特点,目前在机理上并未得到明确的阐释。
值得一提的是,在7月19日,Nature杂志在线发表清华大学李海涛课题组和闫创业课题组题为Diverse modes of H3K36me3-guided nucleosomal deacetylation by Rpd3S的文章,通过结构和生化手段对Rpd3S在H3/H4 deacetylation构象下的分子机制做了详尽的探讨。本研究对前述H3/H4 deacetylation的工作机制做了进一步的验证和支持,同时提出了Rpd3S在不同H3K36me3和linker DNA协作的条件下多个全新的结合模型,发现Rpd3S复合物各亚基的组装模式以及识别核小体底物的关键氨基酸位点,揭示了Rpd3S通过调整与核小体的相对位置实现对不同组蛋白去乙酰化的分子机制;同时,也发现了Rpd3S完成去乙酰化与Hho1发生时空伴随,可能是Rpd3S去乙酰化后移向+1核小体与Hho1协同参与组装和压缩染色质并进一步沉默基因转录。
广州健康院博士生董淑琦、博士后Nadia Rasheed,澳门大学博士后李华东、博士生王美林为本文共同第一作者,广州健康院何俊研究员与澳门大学William Chong Hang Chao教授为共同通讯作者。该研究得到了国家自然科学基金、中国科学院启动基金以及澳门大学、澳门特别行政区科学技术发展基金等的资助。
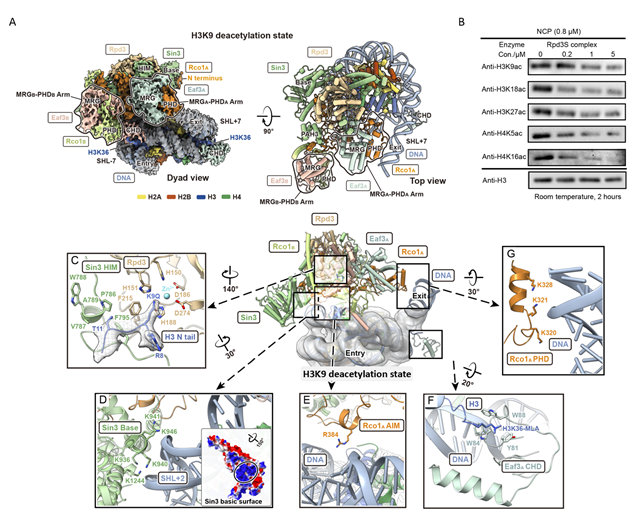
图1. A:Rpd3S去乙酰化H3K9的cryo-EM电子密度图及搭建模型图;B:Rpd3S去乙酰化H3/H4不同赖氨酸残基的western blotting结果;C:H3 N端K9Q与Rpd3的酶活中心相互作用图;D:Sin3 basic surface氨基酸与DNA相互作用图;E:Rco1 AIM与DNA相互作用图;F:CHD芳香笼与H3K36me3相互作用图;G:Rco1 PHD与DNA相互作用图。
附件下载:



