发布时间:2025-07-17
近日,中国科学院广州生物医药与健康研究院王杰课题组与广州国家实验室李亦学课题组合作,成功开发了一种在单细胞水平构建调控网络的新工具ScReNI(Single-cell Regulatory Network Inference)。该工具通过整合单细胞转录组(scRNA-seq)与单细胞染色质可及性(scATAC-seq)数据,实现单细胞精度的基因调控网络推断。相关研究成果以题为“ScReNI: Single-cell Regulatory Network Inference Through Integrating scRNA-seq and scATAC-seq Data”的论文形式发表于国际学术期刊《Genomics, Proteomics & Bioinformatics》。
每个细胞都具有独特的转录组和染色质可及性特征,其内在的基因调控网络也呈现出显著的异质性和细胞特异性。然而,目前仍缺乏能够有效整合单细胞多组学数据,构建单细胞精度调控网络的方法。scRNA-seq和scATAC-seq技术分别能够在单细胞水平上测量基因表达和染色质开放区域,二者的结合为解析精细的细胞特异性调控机制提供了新机会。无论是配对还是未配对的单细胞多组学数据,都为构建单个细胞的调控网络奠定了基础。尽管已有多种方法可用于推断细胞类型特异性的调控网络,但在单细胞尺度上系统构建基因调控关系的研究仍十分有限,特别是在整合scRNA-seq与scATAC-seq数据方面仍面临诸多挑战。
针对上述问题,研究团队提出了一种创新算法ScReNI,能够在单细胞水平上整合scRNA-seq与scATAC-seq数据,实现细胞特异性的调控网络推断。ScReNI的核心设计思路包括以下四个关键步骤(图1):
1. 多组学数据整合:利用Seurat软件中的加权最近邻(weighted nearest neighbor)分析方法,整合配对或未配对的scRNA-seq和scATAC-seq数据;
2. 确定细胞邻域集合:为每个细胞识别k个最近邻细胞,作为后续调控网络推断的基础;
3. 在细胞邻域内建立非线性基因调控关系:采用改进的随机森林模型,结合转录因子(TFs)活性、基因表达和染色质可及性信息,推断非线性调控关系;
4. 识别关键调控因子:基于细胞特异的调控网络,统计识别每个细胞中的富集调控因子,揭示其在不同生物过程中的潜在作用机制。
ScReNI利用邻近细胞的信息模拟局部调控环境,并通过机器学习方法挖掘基因表达与染色质可及性之间的复杂关联,从而构建个性化的调控网络。评估结果显示,ScReNI在调控关系预测和细胞聚类任务中均表现出优异性能,同时还能识别出每个细胞中的关键调控因子,为深入理解单细胞层面的功能调控机制提供了有力支持。
中国科学院广州生物医药与健康研究院的助理研究员徐雪丽、硕士研究生梁嫣然以及博士研究生汤杪庥为本研究的共同第一作者;广州国家实验室李亦学研究员和中国科学院广州生物医药与健康研究院王杰研究员为共同通讯作者。研究工作得到了国家自然科学基金、国家重点研发计划以及广东省科技研发专项的支持。
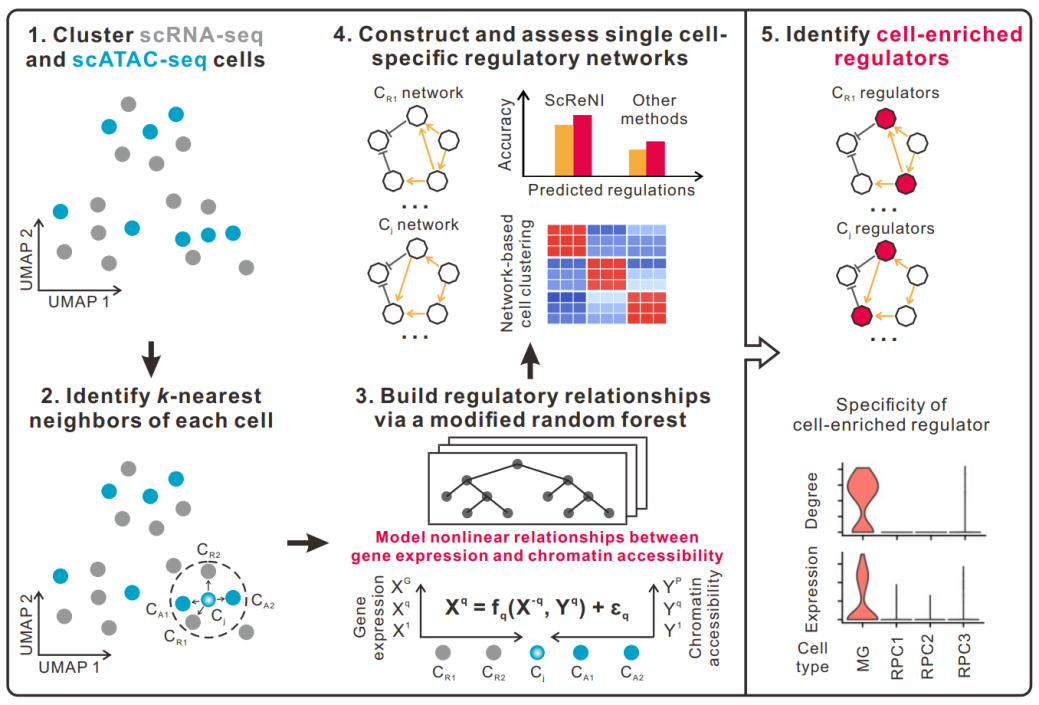
图1 ScReNI流程图
附件下载:



